Bulk SiC system
下文将以 Bulk SiC 系统为例,介绍如何使用 PWMLFF Linear Model 进行训练,以及如何使用训练好的模型进行预测。
整个过程分为以下几个步骤:
1. 产生数据集
以 PWmat AIMD 模拟得到的 SiC 数据为例,数据文件为MOVEMENT,包含 1000 个结构,每个结构包含 216 个原子。
etot.input输入文件示例:
4 1
JOB = MD
MD_DETAIL = 3 1000 1 300 1000
#MD_DETAIL = 3 1000 1 300 300
XCFUNCTIONAL = PBE
Ecut = 50
ECUT2 = 200
MP_N123 = 2 2 2 0 0 0 3
E_ERROR = 0
wg_error = 0
RHO_ERROR = 1E-4
ENERGY_DECOMP = T
IN.ATOM = atom.config
IN.PSP1 = Si.SG15.PBE.UPF
IN.PSP2 = C.SG15.PBE.UPF
- 必选项
ENERGY_DECOMP = T:把总 DFT 能量分解为属于每个原子的能量(原子能量)。结果输出在MOVEMENT文件中。 - 其他参数含义参考PWmat manual。
2. 训练过程
2.1 提取特征
新建目录PWdata,将MOVEMENT文件复制到该目录中。
如果有多个MOVEMENT文件,需要在PWdata目录下分别新建目录(名称任意),并将 MOVEMENT 文件分别放入其中。每个目录只能放一个MOVEMENT。
2.2 训练输入文件
在PWdata同级目录下,新建main.py文件,执行该文件提取特征并进行训练。
输入文件示例:
from PWMLFF.linear_regressor import linear_regressor
if __name__ == "__main__":
# training
# atom type to be used
atom_type = [14,6]
# feature to be used
feature_type = [5]
# create an instance
linReg = linear_regressor(atom_type = atom_type,
feature_type = feature_type,
etot_weight = 0.5,
force_weight = 0.5,
ei_weight = 1.0)
# generate data
# ONLY NEED TO BE DONE ONCE
linReg.generate_data()
# training
linReg.train()
atom_type:原子类型,14 和 6 分别为 Si 和 C 的原子序数feature_type:特征类型,5 对应 Moment Tensor Potential,详见特征类型etot_weight:训练时总能量的权重force_weight:训练时原子力的权重ei_weight:训练时原子能量的权重,设置为 0 时表示不训练原子能量
以下二个函数可根据实际使用情况选择执行:
- linReg.generate_data() - 用于产生特征,仅需运行一次。例如仅需要针对不同的训练权重进行修改,再重新启动训练时,为了节省时间可以注释掉该行再次执行程序。
- linReg.train() - 用于加载特征,对特征进行处理后开始训练。
2.3 运行
以下适用于 Mcloud,提交任务时确保已经加载必要的环境和模块。如conda activate PWMLFF。
#!/bin/sh
#SBATCH --partition=3080ti
#SBATCH --job-name=mlff
#SBATCH --nodes=1
#SBATCH --ntasks-per-node=1
#SBATCH --gres=gpu:1
#SBATCH --gpus-per-task=1
python main.py > log
交互式运行:
$ srun -p 3080ti --gres=gpu:1 --pty /bin/bash
$ python main.py
程序运行后,会在PWdata目录下生成input,output及fread_dfeat目录,同时PWdata目录下会产生新的文件:
-
input
- location
- info_dir
- gen_*_feature.in
-
output
- grid*
-
fread_dfeat
- energyL*
- feat*
- forceL*
- linear*
- weight_feat.*
-
PWdata
- location
- dfeat*
- info*
- trainData*
- inquirepos*
- MOVEMENTall
3. 验证/测试
训练完成后,可以对模型进行验证/测试,以确定模型的拟合效果。
在PWdata同级目录下,新建MD目录,将另一个的MOVEMENT文件复制到该目录中。
该 MOVEMENT 文件同样需要在 AIMD 模拟过程中设置ENERGY_DECOMP = T,以便提取原子能量。
验证输入文件示例:
from PWMLFF.linear_regressor import linear_regressor
if __name__ == "__main__":
# training
# atom type to be used
atom_type = [14,6]
# feature to be used
feature_type = [5]
# create an instance
linReg = linear_regressor(atom_type = atom_type,
feature_type = feature_type,
etot_weight = 0.5,
force_weight = 0.5,
ei_weight = 1.0)
"""
perform evaulation and plot
"""
linReg.evaluate()
linReg.plot_evaluation(plot_elem = False, save_data = False)
- linReg.evaluate() - 该函数调用
/MD/MOVEMENT用于验证训练好的模型 - linReg.plot_evaluation() - 用于绘制验证结果图,结果保存在
plot_data目录下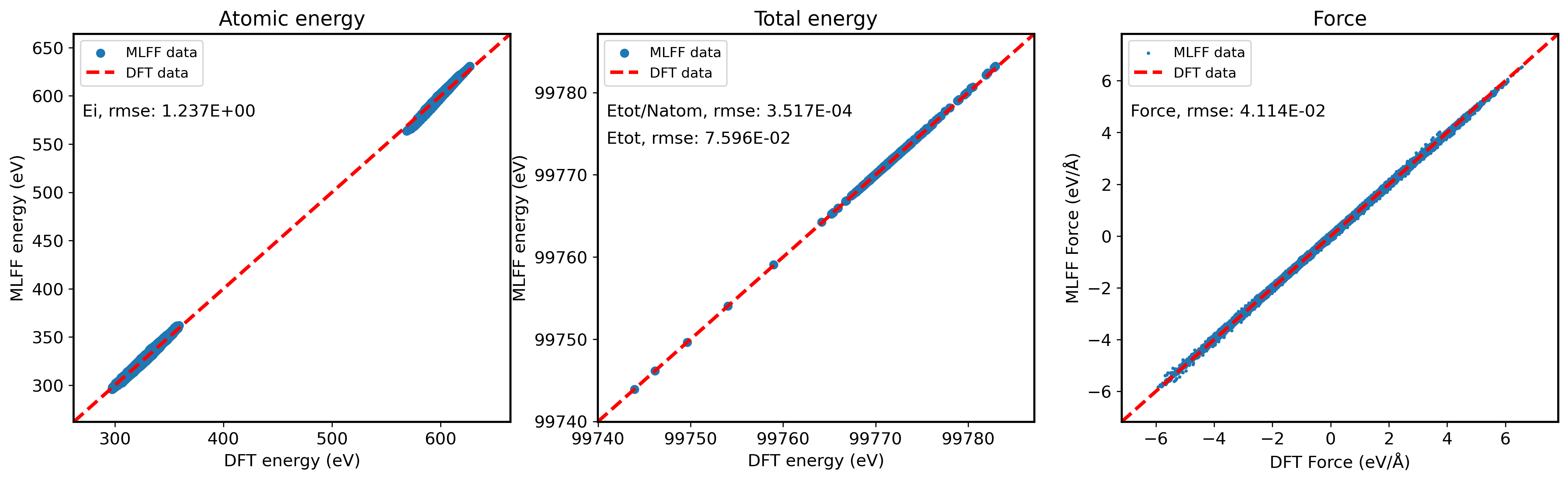
plot_elem: 是否绘制元素能量,默认为False。若为True,则会绘制每种元素的原子能量save_data: 是否保存验证结果,默认为False。
4. 使用模型进行预测
通过对模型的验证,可以确定模型的拟合效果。在模型拟合效果满意的情况下,可以使用模型进行分子动力学预测。
首先需要准备一个�初始构型文件,例如atom.config文件。该文件可以不同于训练时的构型文件,但需要保证原子类型与训练时一致。
输入文件示例:
from PWMLFF.linear_regressor import linear_regressor
if __name__ == "__main__":
# training
# atom type to be used
atom_type = [14,6]
# feature to be used
feature_type = [5]
# create an instance
linReg = linear_regressor(atom_type = atom_type,
feature_type = feature_type,
etot_weight = 0.5,
force_weight = 0.5,
ei_weight = 1.0)
# run MD
# PWmat-style md_detail array
md_detail = [1,1000,1,500,500]
linReg.run_md(md_details = md_detail, follow = False)
- md_detail - 用户必须自行设置的 MD 参数: 1) MD 类型,2) MD 步数,3) MD 步长,4) MD 初始温度,5) MD 终止温度。详细设置请参考PWmat manual
- linReg.run_md() - 运行 AIMD 模拟的函数,该函数运行结束后会在当前目录下生成一个新的
MOVEMENT文件follow: 删除旧的 MOVEMENT 文件,默认为False。若为True,则会在原来的 MOVEMENT 文件上继续追加新的 MD 结果。
5. All-in-one
以下是一个完整的训练、验证、预测的示例(需要准备好atom.config及MD/MOVEMENT):
"""
example of linear fitting workflow
"""
from PWMLFF.linear_regressor import linear_regressor
if __name__ == "__main__":
# training
# atom type to be used
atom_type = [14,6]
# feature to be used
feature_type = [5]
# create an instance
linReg = linear_regressor(atom_type = atom_type,
feature_type = feature_type,
etot_weight = 0.5,
force_weight = 0.5,
ei_weight = 1.0)
# generate data
# ONLY NEED TO BE DONE ONCE
linReg.generate_data()
# training
linReg.train()
"""
perform evaulation and plot
"""
linReg.evaluate()
linReg.plot_evaluation(plot_elem = False, save_data = False)
# run MD
# PWmat-style md_detail array
md_detail = [1,1000,1,500,500]
linReg.run_md(md_details = md_detail, follow = False)